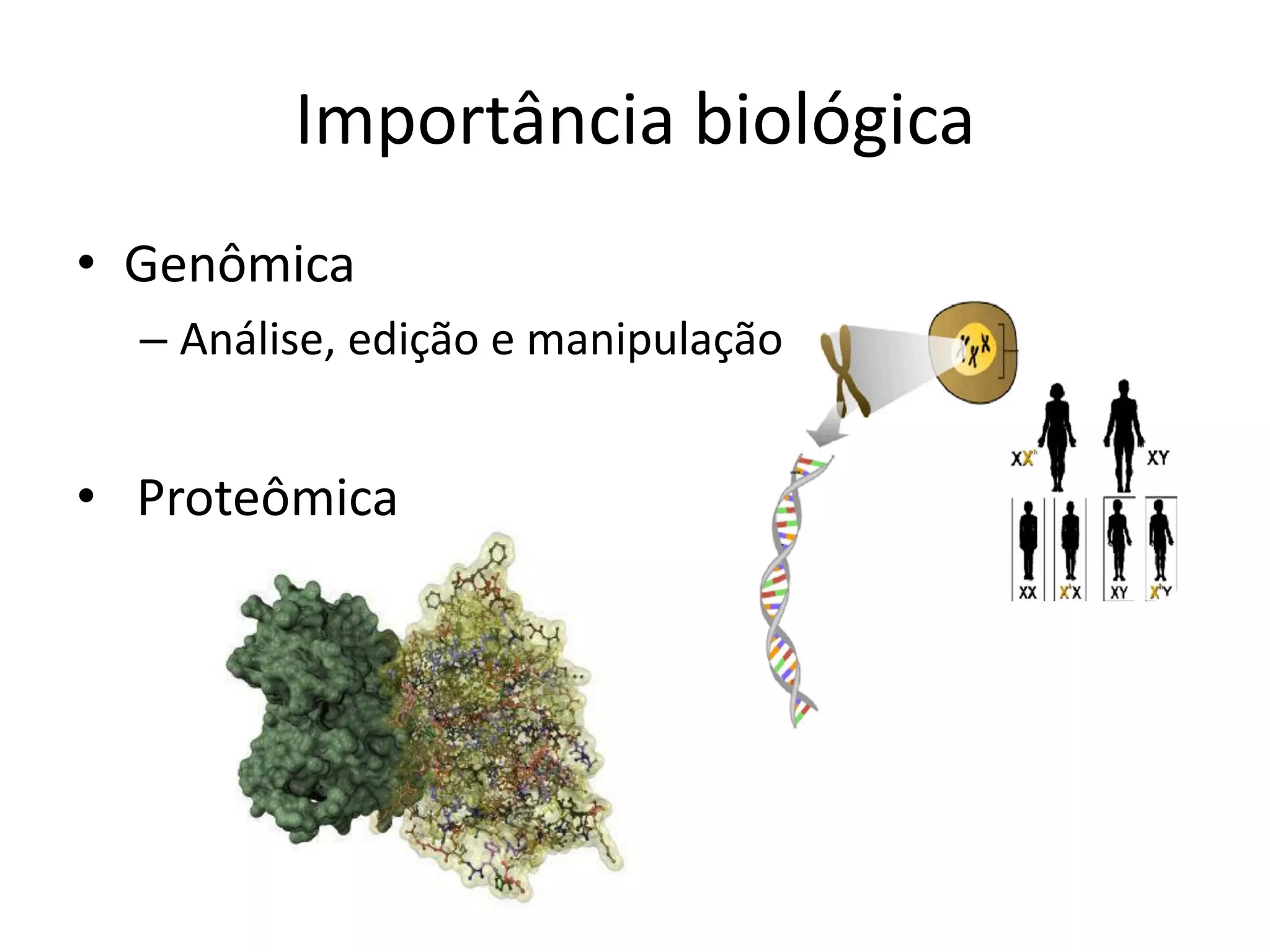
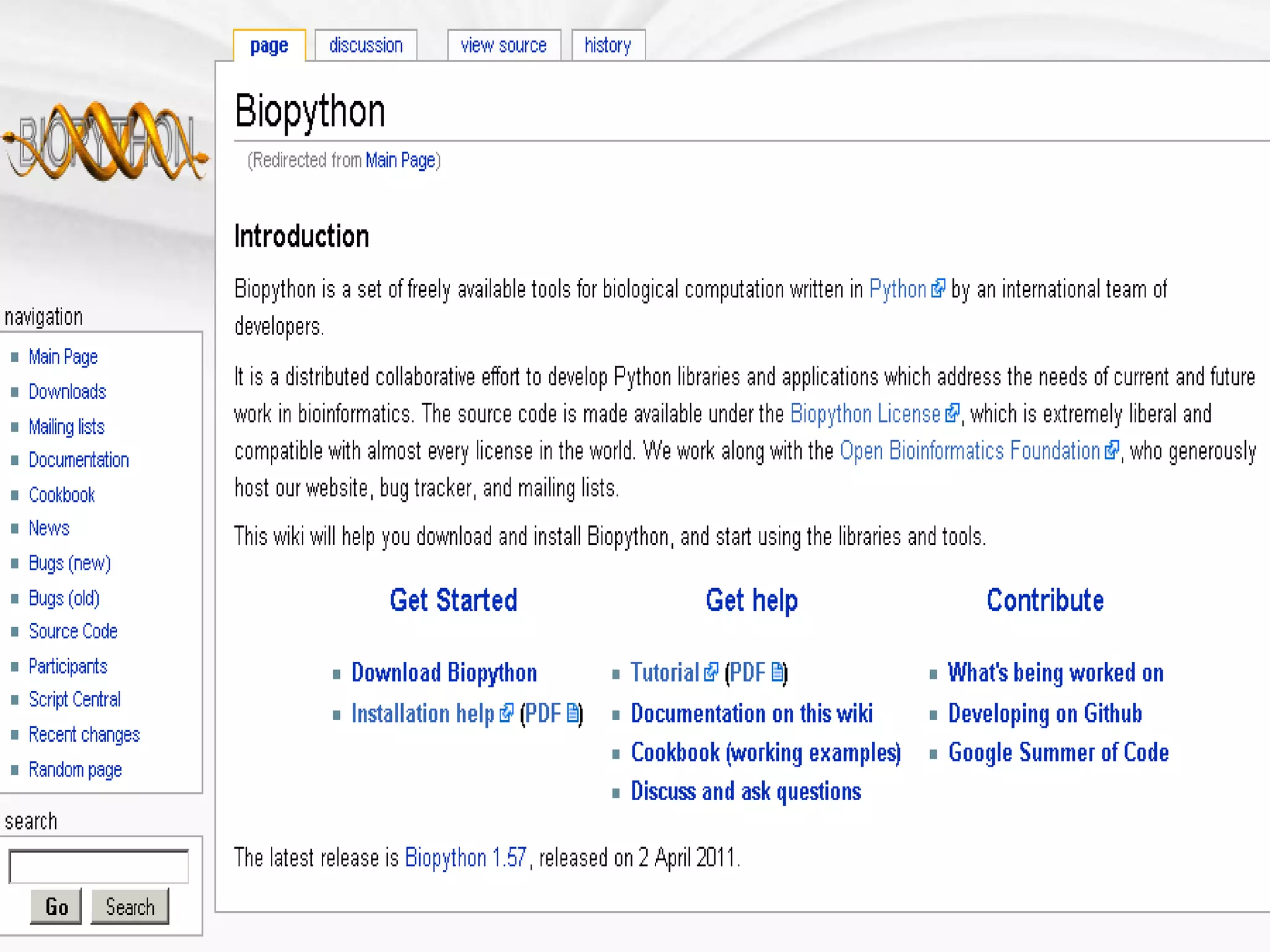
O documento apresenta Péricles Miranda e seu trabalho na área de bioinformática. Ele descreve sua formação acadêmica em engenharia da computação e mestrado em ciência da computação e seu interesse em web, mobile e inteligência artificial. O documento também discute a importância da biologia para a medicina, farmácia e agricultura e como a computação pode ajudar a resolver problemas na biologia. Ele apresenta BioPython como uma biblioteca Python útil para tarefas de bioinformática.















![Na Prática >>> from Bio import SeqIO >>> record = SeqIO.read("NC_005816.gb", "genbank") >>> record SeqRecord(seq=Seq('TGT...CTGTAGA', IUPACDNA()), id='NC_005816.1', name='NC_005816', description='Microtus str. 91001 plasmid pPCP1, dbxrefs=['Project:10638']) Péricles Miranda periclesmiranda@gmail.com](https://image.slidesharecdn.com/biopython-110612192144-phpapp02/75/Bio-python-19-2048.jpg)


![Na Prática • Existe uma série de algoritmos biológicos; • A ideia não é que o usuário os reimplemente; • Existem programas, acessíveis ao BioPython, que realiza o trabalho por você. >>> import Bio.Align.Applications >>> dir(Bio.Align.Applications) ...['ClustalwCommandline', 'DialignCommandline', 'MafftCommandline', 'MuscleCommandline', 'PrankCommandline', 'ProbconsCommandline', 'TCoffeeCommandline' ...] Péricles Miranda periclesmiranda@gmail.com](https://image.slidesharecdn.com/biopython-110612192144-phpapp02/75/Bio-python-22-2048.jpg)


![Na Prática • Buscando o lineage do organismo: >>> from Bio import Entrez >>> handle = Entrez.esearch(db="Taxonomy", term="Cypripedioideae") >>> records[0].keys() [u'Lineage', u'Division', u'ParentTaxId', u'PubDate', u'LineageEx', u'CreateDate', u'TaxId', u'Rank', u'GeneticCode', u'ScientificName', u'MitoGeneticCode', u'UpdateDate'] >>> records[0]["Lineage"] 'cellular organisms; Eukaryota; Viridiplantae; Streptophyta; Streptophytina; Embryophyta; Tracheophyta; Euphyllophyta; Spermatophyta; Magnoliophyta; Liliopsida; Asparagales; Orchidaceae' Péricles Miranda periclesmiranda@gmail.com](https://image.slidesharecdn.com/biopython-110612192144-phpapp02/75/Bio-python-25-2048.jpg)


